Karina Toledo | Agência FAPESP – Um estudo coordenado por pesquisadores da Universidade Estadual de Campinas (Unicamp) e divulgado na plataforma medRxiv traz novas evidências de que o vírus SARS-CoV-2 é capaz de infectar e de se replicar no interior de linfócitos, podendo levar essas células de defesa à morte e comprometer ao menos temporariamente o sistema imunológico.
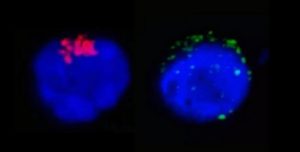
Na imagem da esquerda é possível observar a enzima RdRp do SARS-CoV-2 (em vermelho), que regula a replicação viral, no interior do linfócito T CD4.Na imagem da direita, as manchas verdes representam as proteínas de espícula do SARS-CoV-2 (spike proteins) próximas à membrana da célula de defesa humana (imagem: Henrique Marques-Souza/Unicamp)
Segundo os autores, o novo coronavírus atua por um mecanismo semelhante ao do HIV, causador da Aids. Os dois afetam um tipo de linfócito conhecido como T CD4, que é responsável por coordenar a chamada resposta imune adaptativa – auxiliando tanto os linfócitos B a produzirem anticorpos como os linfócitos T CD8 – responsáveis por reconhecer e matar células infectadas – a se proliferarem. Essa coordenação se dá por meio da liberação de moléculas sinalizadoras conhecidas como citocinas.
“Nossos resultados sugerem que, em alguns pacientes, o novo coronavírus pode causar um quadro de imunodeficiência aguda não apenas porque mata parte dos linfócitos T CD4, mas também porque prejudica a função dessas células. Isso faz com que os linfócitos T CD8 se proliferem menos e os linfócitos B produzam anticorpos com menor afinidade e duração. Seria um efeito semelhante ao do HIV, só que agudo”, explica à Agência FAPESP Alessandro Farias, chefe do Departamento de Genética, Evolução, Microbiologia e Imunologia do Instituto de Biologia (IB) da Unicamp, que coordenou a investigação ao lado do professor Marcelo Mori.
As conclusões do artigo, ainda em processo de revisão por pares, estão baseadas principalmente em experimentos com culturas primárias de linfócitos (isolados do sangue de voluntários não infectados e de pacientes com COVID-19) conduzidos no Laboratório de Estudos de Vírus Emergentes (Leve) do IB-Unicamp, com apoio da FAPESP.
Na primeira etapa da pesquisa, os pesquisadores incubaram células de doadores saudáveis com o SARS-CoV-2 e acompanharam o que acontecia nas 24 horas seguintes por meio de diferentes técnicas, como hibridização in situ, microscopia eletrônica de transmissão e RT-PCR (a mesma usada para diagnosticar a infecção na fase aguda).
“Fizemos esse ensaio apenas com linfócitos T CD4 e T CD8 por serem os tipos mais diminuídos nos pacientes com COVID-19 grave. As análises confirmaram a presença do novo coronavírus no interior de aproximadamente 40% dos T CD4, sendo que 10% dessas células morreram ao final do período de observação. Os linfócitos T CD8 não foram afetados”, conta Farias.
Os pesquisadores observaram ainda que a carga viral mais do que dobrou entre as medições feitas duas e 24 horas após o início do teste – sinal de que o vírus estava se replicando nas células em cultura.
O passo seguinte foi analisar com as mesmas ferramentas os linfócitos T CD4 isolados de pacientes diagnosticados com COVID-19 em busca de sinais do SARS-CoV-2. Nas pessoas que apresentavam quadros moderados da doença foram encontrados poucos linfócitos infectados e eles estavam produzindo, como era esperado, a citocina interferon-gama (IFN-γ) – importante para a resposta antiviral. Já nos pacientes graves, além de haver um número muito maior de linfócitos com o vírus, as células estavam produzindo no lugar da IFN-γ a interleucina-10 (IL-10), uma citocina com ação anti-inflamatória. Ou seja, nesses doentes com COVID-19 severa, os linfócitos T CD4 estavam sinalizando para o sistema imune a necessidade de frear o combate ao vírus.
Segundo Farias, isso explicaria por que muitas pessoas nessa condição apresentam alterações na resposta imune adaptativa (aquela que é específica para cada patógeno), como linfopenia (redução na concentração geral de linfócitos no sangue), exaustão de células T e produção comprometida de anticorpos.
“A produção de IL-10 desliga o sistema imune e permite ao vírus permanecer mais tempo no organismo. Por enquanto ainda não é possível saber o que é causa e o que é consequência, ou seja, se esses pacientes evoluíram para a forma grave porque tinham mais linfócitos T CD4 infectados ou o contrário. Mas há uma clara associação entre esses dois fatores”, afirma Farias.
Abrindo passagem
Vários estudos já publicados apontam a molécula ACE2 (enzima conversora de angiotensina 2, na sigla em inglês) como a principal porta de entrada para o SARS-CoV-2 na superfície das células humanas. No entanto, os linfócitos T CD4 sabidamente expressam uma quantidade muito pequena dessa enzima na superfície de sua membrana plasmática, que é recoberta pela proteína que dá nome à célula: a CD4.
Para desvendar a estratégia usada pelo novo coronavírus para entrar nesses linfócitos – que normalmente são refratários à infecção por vírus e bactérias – o grupo da Unicamp realizou dois novos testes com as amostras de doadores saudáveis. No primeiro, antes de colocar o vírus, foram acrescentados na cultura celular anticorpos capazes de neutralizar a proteína CD4. No segundo experimento, foram colocados anticorpos contra a ACE2.
“Nossa hipótese era de que o SARS-CoV-2 conseguiria entrar na célula usando apenas a CD4, mas quando neutralizamos também a ACE2 a infecção foi totalmente bloqueada. Isso mostra que, mesmo em pequena quantidade, a ACE2 é necessária para a invasão do linfócito”, diz Farias.
Ensaios de interação entre moléculas in vitro revelaram que a proteína de espícula do SARS-CoV-2, aquela que forma a coroa presente na superfície viral, é capaz de se ligar à CD4 do linfócito.
“Acreditamos que o vírus apresenta uma artimanha para entrar nessa célula. Ele usa a proteína CD4 apenas para ficar perto da membrana celular e conseguir localizar a ACE2, que então lhe dá passagem para o meio intracelular”, explica o pesquisador.
Na terceira e última etapa da pesquisa, feita em parceria com o professor da Universidade de São Paulo (USP) Helder Nakaya, os pesquisadores usaram técnicas de bioinformática para reanalisar dados de um estudo publicado por cientistas chineses em maio, na revista Nature Medicine, no qual foi feito o sequenciamento de leucócitos isolados da secreção pulmonar de pacientes com COVID-19 grave por uma técnica conhecida como single-cell sequencing.
“O algoritmo desenvolvido pelo grupo de Nakaya permitiu identificar o genoma viral também nos linfócitos que estavam no pulmão dos pacientes, trazendo um novo nível de evidência e ainda mais confiabilidade para os achados”, afirma Farias.
A pesquisa contou também com a colaboração de pesquisadores da Universidade Estadual Paulista (Unesp), do Centro Nacional de Pesquisa em Energia e Materiais (CNPEM), da Fundação Oswaldo Cruz (Fiocruz) e do Instituto D’Or de Pesquisa e Ensino (Idor). O apoio da FAPESP se deu por meio de diversos auxílios e bolsas (19/16116-4, 19/06372-3, 20/04583-4, 13/08293-7, 20/04579-7, 15/15626-8, 18/14933-2, 20/04746-0, 19/00098-7, 20/04919-2, 17/01184-9, 19/17007-4, 19/22398-2, 19/05155-9, 19/06459-1, 19/04726-2, 17/23920-9, 16/24163-4 e 16/23328-0).
No momento, o grupo tenta detalhar ainda mais os efeitos causados pela entrada do SARS-CoV-2 no linfócito T CD4. O objetivo é encontrar formas de intervir nesse processo, o que poderia em tese ajudar a combater a infecção.
“Já temos linfócitos isolados de mais de 350 pacientes que pretendemos usar nos experimentos em laboratório e também vamos fazer testes com camundongos geneticamente modificados para expressar a ACE2 humana. Uma das ideias é avaliar o efeito de moléculas capazes de inibir a interação entre a proteína de espícula do vírus e a CD4”, adianta o pesquisador.
O artigo SARS-CoV-2 Uses CD4 to Infect T Helper Lymphocytes pode ser lido em www.medrxiv.org/content/10.1101/2020.09.25.20200329v1.full.pdf.

